Import von .txt und .csv
Als erstes werden wir globale Datensatz-Dateiformate importieren. Dies sind .txt und .csv. Beim Format .csv gibt es wieder eine Sprachenbesonderheit. Im Standard (englische Sprachversion am Computer) ist das Trennzeichen der Daten ein , (Komma), auf Computern mit deutscher Spracheinstellung weicht dies ab und das Datentrennzeichen ist ein ; (Semikolon).
Import einer .txt-Datei
Eine .txt-Datei ist eine reine Textdatei in der Daten mit einem Tabulator getrennt sind (siehe Screenshot). Auch hier ist das Format in der Konvention, dass in der Spalte Variablen eingetragen sind und in den Zeilen die Fälle. In der Regel stellt die erste Zeile keinen Fall dar, sondern beinhaltet wie im Beispiel die Variablennamen.
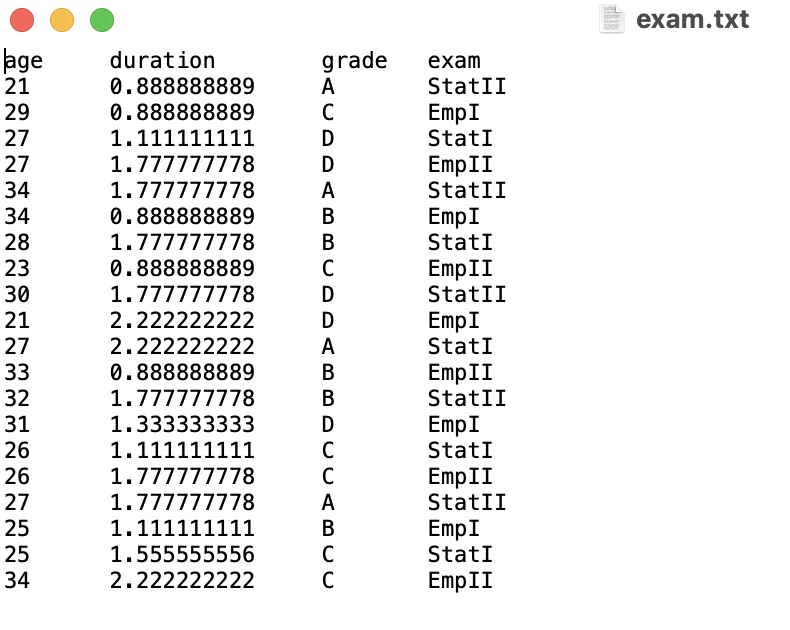 Im Beispiel haben wir also einen Datensatz mit vier Variablen:
Im Beispiel haben wir also einen Datensatz mit vier Variablen: age, duration, grade und exam.
Wenn du Dateien lädtst, musst du dir in Erinnerung rufen, was ein Dateipfad ist. Wenn du das nicht mehr weißt, geh zurück in den Lernblock 1. RStudio Cloud erleichtert uns dies, indem der Pfad sehr leicht angewählt werden kann: In den RStudio Projekte ist immer ein Ordner namens data vorhanden, der wie folgt angesteuert wird: ./data/ und nach dem zweiten Slash folgt der Dateiname. Wie es bei einer lokalen Installation funktioniert, kannst du ganz unten nachlesen. Das ist aber für den Kurs erstmal nicht relevant, da wir alle mit der RStudio Cloud arbeiten.
In R ist das Environment der Ort, wo wir alle geladenen und gespeicherten Daten und Objekte sehen.
Um nun die .txt-Datei (tab-separated file) in R zu laden, benötigen wir die Funktion read.table(). Immer wenn man Hilfe zur einer bekannten Funktion benötigt, kann man einfach ein ? vor diese setzen und die Argumente leer lassen. Im Help-Tab in der Files-Kachel öffnen sich dann Hinweise zur Funktion.
Probieren wir es aus.
# Falls man Hilfe zur Funktion benötigt:
?read.table()Jetzt wollen wir die Daten in R importieren, damit wir mit diesen arbeiten können. Die Datensätze sind bereits im Ordner data gespeichert. Datensatz-Objekte benennt man am besten so, dass man intuitiv darauf schließen kann, um welche Daten es sich handelt. Innerhalb der Import-Funktion (read.table()) gibst du einfach den gesamten Pfad zur Datei an. Da die erste Zeile in der Datei die Variablennamen beinhaltet, geben wir im letzten Argument header = TRUE an, dass dort die Variablennamen zu finden sind. Das Argument sep = "\t" gibt einfach an, dass das Trennzeichen für die Daten ein Tabulator ist.
exam <- read.table(
"./data/exam.txt",
sep = "\t",
header = TRUE
) Wir haben nun die Daten importiert und einen neuen data frame importiert, der exam heißt! Dieser ist im Environment bei dir nun sichtbar.
Struktur eines Datensatzes
Es gibt eine Reihe von Helfer-Funktionen, um einen Überblick über importierte Daten zu bekommen. Diese wirst du jetzt kennenlernen.
Um die Struktur des Datensatzes anzeigen zu lassen, ruft man die folgende Funktion auf:
str(exam)## 'data.frame': 20 obs. of 4 variables:
## $ age : int 21 29 27 27 34 34 28 23 30 21 ...
## $ duration: num 0.889 0.889 1.111 1.778 1.778 ...
## $ grade : chr "A" "C" "D" "D" ...
## $ exam : chr "StatII" "EmpI" "StatI" "EmpII" ...Mit der Funktion head() können wir einen ersten Einblick in die Daten bekommen (die ersten \(6\) Fälle):
head(exam)## age duration grade exam
## 1 21 0.8888889 A StatII
## 2 29 0.8888889 C EmpI
## 3 27 1.1111111 D StatI
## 4 27 1.7777778 D EmpII
## 5 34 1.7777778 A StatII
## 6 34 0.8888889 B EmpIWir können uns auch mehr als \(6\) Fälle anzeigen lassen, dazu fügt man ein zweites Argument hinzu, in dem die Anzahl genannt wird.
head(
exam,
n = 10 # hier kann die Anzahl verändert werden
)## age duration grade exam
## 1 21 0.8888889 A StatII
## 2 29 0.8888889 C EmpI
## 3 27 1.1111111 D StatI
## 4 27 1.7777778 D EmpII
## 5 34 1.7777778 A StatII
## 6 34 0.8888889 B EmpI
## 7 28 1.7777778 B StatI
## 8 23 0.8888889 C EmpII
## 9 30 1.7777778 D StatII
## 10 21 2.2222222 D EmpIWir können auch einzelne Variablen innerhalb des Datensatzes ansprechen. Dazu nennen wir den Datensatz und adressieren mit dem $ eine einzelne Variable:
head(exam$grade)## [1] "A" "C" "D" "D" "A" "B"So jetzt hast du das geschafft und die Anwendung auf andere Dateiformate ist gar nicht so viel mehr Arbeit. Importieren wir als nächstes eine .csv-Datei.
Import einer .csv-file
Um eine .csv-file zu laden, benötigen wir kein weitere library. Die Funktion (wie die oben) ist in R-Base enthalten.
Die Datei inkludiert den gleichen Datensatz, nur als .csv-Datei.
Wir benötigen die Funktion read.csv() bzw. read.csv2(). Dies hängt davon ab, ob ihr die .csv-Datei bereits einmal auf eurem Rechner (Excel oder LibreOffice) geöffnet habt. Wenn ja und eure Computersprache Deutsch ist, müsst ihr read.csv2() verwenden. Habt ihr die Datei nicht ein einziges Mal geöffnet oder englische Spracheinstellungen nutzt read.csv().
Probieren wir es mal aus!
examcsv <- read.csv(
"./data/exam.csv",
header = TRUE
)
examcsv2 <- read.csv2(
"./data/exam.csv",
header = TRUE
)Hier seht ihr einen Screenshot der zwei importierten Datensätze mit den unterschiedlichen Funktionen.
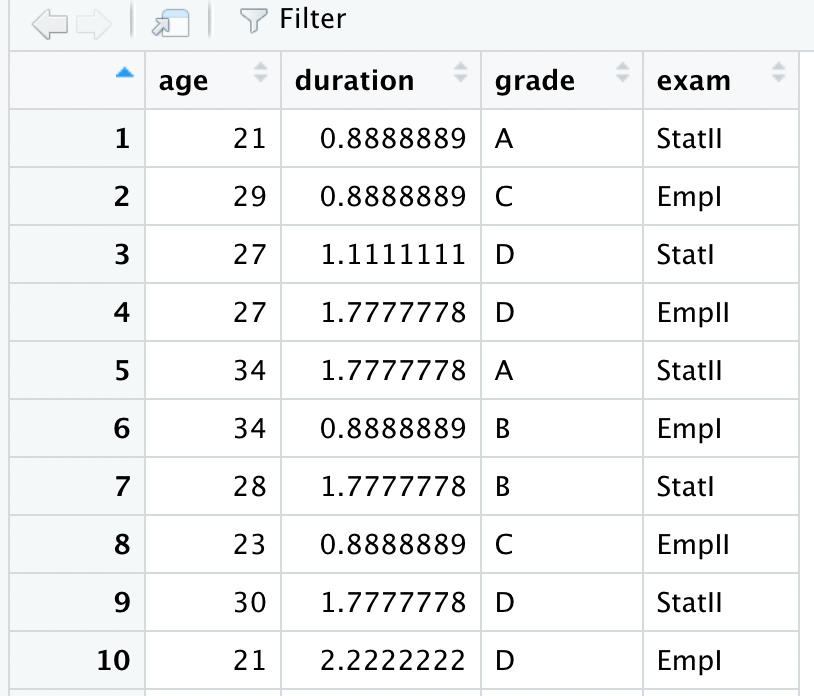
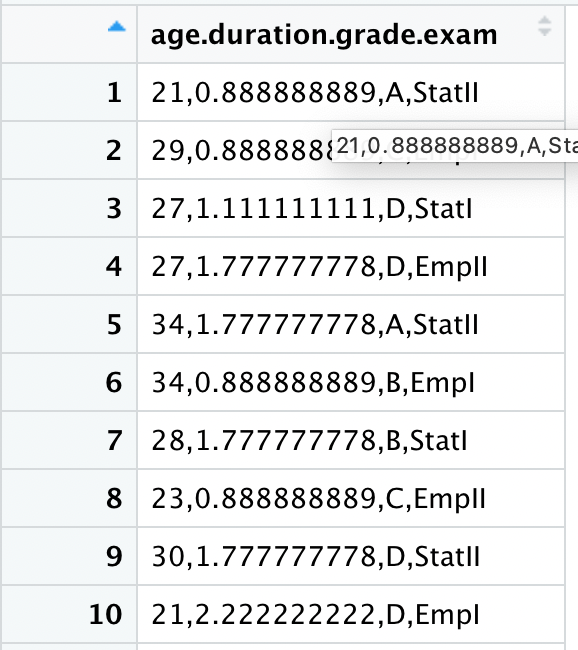
Es ist wichtig zu verstehen, welches Dateiformat vorliegt und wie dies zu importieren ist, denn so können Fehler beim Import leicht vermieden werden.
Gehen wir nun über in die Formate, die R zur Verfügung stellt!
Lokales Arbeiten
Wenn du mal später lokal arbeitest, musst du immer den direkten Pfad der Datei angeben. Oder du speicherst in einem Objekt den Pfad und nutzt dann dieses Objekt innerhalb der Helfer-Funktion file.path(). Das hat den Vorteil, dass du nicht ständig den Pfad eingeben musst, denn es empfiehlt es alle Datensätze an einem Ort zu speichern. Mit diesem einen zentralen Ort, kannst du einfach ein Objekt erstellen, dass diesen Pfad als Text beinhaltet. Du erstellst also wie im Lernblock 1 einfach ein Objekt, dass character beinhaltet, die hier sinnvoll den Pfad angeben. So muss der Pfad nicht immer neu eingegeben werden.
Wichtig: Der Pfad geht nur bis zum Ordner, in dem die Datei liegt. Die Datei wird nicht mit in das path-Objekt gespeichert.
Lege also ein Objekt an, dass den Pfad beinhaltet. Wir nennen dies in der Regel path.
path <- "C:/Dateipfad zum gespeicherten Objekt/"
# kopiere dies aus dem Explorer oder Finder und denke daran bei Windows die Backslashes zu Slashes zu ändernIn der Import-Funktion ergänzt du einfach die Helfer-Funktion file.path(), deren erstes Argument das Objekt path (der Character zu deinem Objekt) und deren zweites Argument der Dateiname ist (hier exam.txt). Alles andere bleibt wie oben!
exam <- read.table(
file.path(
path,
"exam.txt"
),
sep = "\t",
header = TRUE
)